Мерцание - GLIMMER
| Разработчики) | Стивен Зальцберг и Артур Делчер |
|---|---|
| Стабильный выпуск | 3.02 / 9 мая 2006 г. |
| Доступно в | C ++ |
| Тип | Биоинформатика орудие труда |
| Лицензия | Сертифицированное OSI программное обеспечение с открытым исходным кодом под лицензией Artistic License |
| Интернет сайт | ccb |
В биоинформатика, GLIMMER (локатор генов и интерполированная марковская модель ER) используется, чтобы найти гены в прокариотических ДНК.[1] "Он эффективен для поиска генов в бактерии, архея, вирусы, обычно находя 98-99% всего относительно долгого гены, кодирующие белок ".[1] GLIMMER была первой системой, в которой использовался интерполированный Марковская модель [2] для идентификации кодирующих областей. Программное обеспечение GLIMMER имеет открытый исходный код и поддерживается Стивен Зальцберг, Art Delcher и их коллеги из Центр вычислительной биологии[3] в Университет Джона Хопкинса. Оригинальные алгоритмы и программное обеспечение GLIMMER были разработаны Артом Делчером, Саймоном Касифом и Стивеном Зальцбергом и применены к аннотации бактериального генома в сотрудничестве с Оуэн Уайт.
Версии
GLIMMER 1.0
Первая версия GLIMMER, т.е. GLIMMER 1.0, была выпущена в 1998 году и опубликована в газете. Идентификация микробных генов с использованием интерполированной марковской модели.[1] Марковские модели использовались для идентификации микробных генов в GLIMMER 1.0. GLIMMER учитывает локальные зависимости последовательности композиций, что делает GLIMMER более гибким и мощным по сравнению с фиксированным порядком. Марковская модель.
Было проведено сравнение между интерполированный Марковская модель, используемая GLIMMER, и марковская модель пятого порядка в статье Идентификация микробных генов с использованием интерполированных марковских моделей.[1] "Алгоритм GLIMMER обнаружил 1680 генов из 1717 аннотированных генов в Haemophilus influenzae где пятый порядок Марковская модель найдено 1574 гена. GLIMMER обнаружил 209 дополнительных генов, которые не были включены в 1717 аннотированных генов, где пятый порядок Марковская модель найдено 104 гена ".[1]
GLIMMER 2.0
Вторая версия GLIMMER, то есть GLIMMER 2.0, была выпущена в 1999 году и опубликована в газете. Улучшенная идентификация микробов с помощью GLIMMER.[4] Эта бумага[4] обеспечивает значительные технические улучшения, такие как использование интерполированной модели контекста вместо интерполированной модели Маркова и разрешение перекрывающихся генов, что повышает точность GLIMMER.
Интерполированный контекстные модели используются вместо интерполированный Марковская модель, дающая возможность выбора любой базы. В интерполированной марковской модели распределение вероятностей базы определяется из непосредственно предшествующих баз. Если непосредственно предшествующая база не имеет значения аминокислота трансляция, интерполированная марковская модель по-прежнему учитывает предыдущую базу для определения вероятности данной базы, тогда как интерполированная контекстная модель, которая использовалась в GLIMMER 2.0, может игнорировать нерелевантные базы. Количество ложноположительных прогнозов было увеличено в GLIMMER 2.0, чтобы уменьшить количество ложноотрицательных прогнозов. Перекрывающиеся гены также разрешены в GLIMMER 2.0.
В статье были сделаны различные сравнения между GLIMMER 1.0 и GLIMMER 2.0. Улучшенная идентификация микробов с помощью GLIMMER[4] что показывает улучшение в более поздней версии. «Чувствительность GLIMMER 1.0 колеблется от 98,4 до 99,7% со средним значением 99,1%, тогда как GLIMMER 2.0 имеет диапазон чувствительности от 98,6 до 99,8% со средним значением 99,3%. GLIMMER 2.0 очень эффективен в обнаружении генов высокой плотности. паразит Trypanosoma brucei, ответственный за причинение Африканская сонная болезнь идентифицируется GLIMMER 2.0 " [4]
GLIMMER 3.0
Третья версия GLIMMER, «GLIMMER 3.0» была выпущена в 2007 году и опубликована в газете. Идентификация бактериальных генов и ДНК эндосимбионтов с помощью Glimmer.[5] В этом документе описывается несколько основных изменений, внесенных в систему GLIMMER, включая улучшенные методы идентификации кодирующих областей и запуска кодон. Подсчет ORF в GLIMMER 3.0 выполняется в обратном порядке, то есть начиная со стоп-кодона и возвращается к стартовому кодону. Обратное сканирование помогает более точно идентифицировать кодирующую часть гена, которая содержится в контекстном окне IMM. GLIMMER 3.0 также улучшает сгенерированные данные обучающего набора, сравнивая длинную ORF с универсальным распределением аминокислот в сильно различающихся бактериальных геномах. GLIMMER 3.0 имеет средний выход длинной ORF 57% для различных организмов, тогда как GLIMMER 2.0 имеет средний длинный -ORF выход 39% ».[5]
GLIMMER 3.0 снижает частоту ложноположительных прогнозов, которые были увеличены в GLIMMER 2.0, чтобы уменьшить количество ложноотрицательных прогнозов. «GLIMMER 3.0 имеет точность предсказания начальной точки 99,5% для совпадений 3'5 ', тогда как GLIMMER 2.0 имеет 99,1% для совпадений 3'5'. GLIMMER 3.0 использует новый алгоритм для сканирования областей кодирования, новый модуль обнаружения начальных сайтов и архитектура, которая объединяет все прогнозы генов по всему геному ".[5]
Теоретико-биологический фундамент
Проект GLIMMER помог ввести и популяризировать использование моделей переменной длины в вычислительной биологии и биоинформатике, которые впоследствии были применены к многочисленным задачам, таким как классификация белков и другие. Первоначально моделирование переменной длины было предложено теоретиками информации, а затем гениально применено и популяризировано в области сжатия данных (например, сжатие Зива-Лемпеля). Прогнозирование и сжатие тесно связаны с использованием Минимальная длина описания Принципы. Основная идея - создать словарь часто употребляемых слов (мотивов в биологических последовательностях). Интуиция подсказывает, что часто встречающиеся мотивы могут быть наиболее полезными и информативными. В GLIMMER интерполированная модель представляет собой смесь вероятностей этих относительно общих мотивов. Подобно развитию HMM в вычислительной биологии, на авторов GLIMMER концептуально повлияло предыдущее применение другого варианта интерполированных моделей Маркова для распознавания речи такими исследователями, как Фред Елинек (IBM) и Эрик Ристад (Принстон). Алгоритм обучения в GLIMMER отличается от этих более ранних подходов.
Доступ
GLIMMER можно скачать с Домашняя страница Glimmer (требуется C ++ компилятор В качестве альтернативы онлайн-версия размещается на NCBI [1].
Как это устроено
- GLIMMER в первую очередь ищет долго-ORFS. Открытая рамка считывания может перекрываться с любой другой открытой рамкой считывания, которая будет разрешена с использованием техники, описанной в подразделе. Используя эти длинные ORFS и следуя определенному распределению аминокислот, GLIMMER генерирует Обучающий набор данные.
- Используя эти обучающие данные, GLIMMER обучает все шесть марковских моделей кодирования ДНК от нулевого до восьмого порядка, а также обучает модель для некодирующая ДНК
- GLIMMER пытается вычислить вероятности на основе данных. Основываясь на количестве наблюдений, GLIMMER определяет, следует ли использовать фиксированный порядок Марковская модель или интерполированный Марковская модель.
- Если количество наблюдений превышает 400, GLIMMER использует марковскую модель фиксированного порядка для получения этих вероятностей.
- Если количество наблюдений меньше 400, GLIMMER использует интерполированный Модель Маркова, которая кратко объясняется в следующем подразделе.
- GLIMMER получает оценку для каждой длинной ORF, созданной с использованием всех шести моделей кодирующей ДНК, а также с использованием модели некодирующей ДНК.
- Если оценка, полученная на предыдущем этапе, превышает определенный порог, GLIMMER предсказывает, что это ген.
Шаги, описанные выше, описывают основные функции GLIMMER. В GLIMMER внесены различные улучшения, некоторые из которых описаны в следующих подразделах.
Система GLIMMER
Система GLIMMER состоит из двух программ. Первая программа под названием build-imm, которая принимает входной набор последовательностей и выводит интерполированный Марковская модель выглядит следующим образом.
Вероятность для каждой базы, то есть A, C, G, T для всех k-mers для 0 ≤ k ≤ 8 вычисляется. Затем для каждого к-мер, GLIMMER вычисляет вес. Вероятность новой последовательности вычисляется следующим образом.
где n - длина последовательности это олигомер в позиции x. , то -порядок интерполированный Оценка марковской модели рассчитывается как
"куда это вес к-мер в позиции x-1 в последовательности S и - оценка вероятности того, что база находится в позиции x в -Заказать модель ".[1]
Вероятность основания с учетом i предыдущих баз вычисляется следующим образом.
"Значение связана с можно рассматривать как меру уверенности в точности этого значения как оценку истинной вероятности. GLIMMER использует два критерия для определения . Первый из них - это простое частотное вхождение, в котором количество вхождений контекстной строки в обучающих данных превышает определенное пороговое значение, тогда установлен на 1.0. Текущее значение по умолчанию для порога 400, что дает 95% уверенности. Если количество экземпляров контекстной строки недостаточно, build-imm использует дополнительные критерии для определения ценить. Для данной контекстной строки длины i, build-imm сравнивает наблюдаемые частоты следующей базы , , , с предварительно рассчитанным интерполированный Вероятности марковской модели с использованием следующего более короткого контекста, , , , . Используя test, build-imm определяет, насколько вероятно, что четыре наблюдаемые частоты согласуются со значениями IMM из следующего более короткого контекста ".[1]
Вторая программа, называемая glimmer, затем использует этот IMM для идентификации предполагаемого гена во всем геноме. GLIMMER идентифицирует все открытая рамка чтения которые набирают больше порогового значения и проверяют перекрывающиеся гены. Устранение перекрывающихся генов объясняется в следующем подразделе.
Уравнения и объяснение используемых выше терминов взяты из статьи «Идентификация микробных генов с использованием интерполированных марковских моделей.[1]
Устранение перекрывающихся генов
В GLIMMER 1.0, когда два гена A и B перекрываются, оценивается область перекрытия. Если A длиннее, чем B, и если A имеет более высокие оценки в области перекрытия, и если перемещение начального сайта B не устраняет перекрытие, то B отклоняется.
GLIMMER 2.0 предоставил лучшее решение для устранения перекрытия. В GLIMMER 2.0, когда два потенциальных гена A и B перекрываются, область перекрытия оценивается. Предположим, ген А набрал больше баллов, рассматриваются четыре разные ориентации.
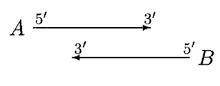
В приведенном выше случае перемещение начальных участков не устраняет перекрытие. Если A значительно длиннее, чем B, то B отвергается, иначе оба A и B называются генами с сомнительным перекрытием.
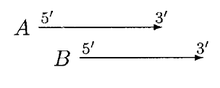
В приведенном выше случае перемещение B может устранить перекрытие, A и B можно назвать неперекрывающимися генами, но если B значительно короче, чем A, то B отклоняется.

В приведенном выше случае перемещение A может устранить перекрытие. A перемещается только в том случае, если перекрытие составляет небольшую долю от A, иначе B отклоняется.

В приведенном выше случае можно перемещать как A, так и B. Сначала мы перемещаем начало B до тех пор, пока область перекрытия не наберет более высокий балл для B. Затем мы перемещаем начало A, пока оно не станет более высоким. Затем снова B и так далее, пока либо перекрытие не будет устранено, либо дальнейшие ходы не будут выполнены.
Приведенный выше пример был взят из статьи «Идентификация бактериальных генов и ДНК эндосимбионтов с помощью Glimmer».[5]
Сайты связывания рибосом
Сайт связывания рибосомы (RBS) сигнал может быть использован для определения истинного положения начального участка. Результаты GLIMMER передаются в качестве входных данных для программы RBSfinder для прогнозирования сайтов связывания рибосом. GLIMMER 3.0 интегрирует программу RBSfinder в функцию прогнозирования генов.
Программное обеспечение ELPH (которое было определено как высокоэффективное при идентификации RBS в документе[5]) используется для идентификации RBS и доступен по этому адресу интернет сайт. Выборка Гиббса алгоритм используется для определения общих мотив в любом наборе последовательностей. Это общий мотив последовательности и их длина вводятся в ELPH. Затем ELPH вычисляет матрицу весов положения (PWM), которая будет использоваться GLIMMER 3 для оценки любого потенциального RBS, обнаруженного RBSfinder. Вышеупомянутый процесс выполняется, когда у нас есть значительное количество обучающих генов. Если количество обучающих генов недостаточное, GLIMMER 3 может сам настроить себя для генерации набора прогнозов генов, которые можно использовать в качестве входных данных для ELPH. ELPH теперь вычисляет ШИМ, и этот ШИМ можно снова использовать на том же наборе генов, чтобы получить более точные результаты для стартовых сайтов. Этот процесс можно повторять для многих итераций, чтобы получить более согласованные результаты ШИМ и прогнозирования генов.
Спектакль
Glimmer поддерживает усилия по аннотации генома для широкого спектра бактериальных, архейных и вирусных видов. В крупномасштабных усилиях по обновлению аннотаций в Банке данных ДНК Японии (DDBJ, который отражает Генбанк ). Kosuge и другие. (2006)[6] изучили методы поиска генов, используемые для 183 геномов. Они сообщили, что из этих проектов Глиммер был генофондом для 49%, за которым следовали GeneMark с 12%, с другими алгоритмами, используемыми в 3% или меньше проектов. (Они также сообщили, что 33% геномов использовали «другие» программы, что во многих случаях означало, что они не могли идентифицировать метод. За исключением этих случаев, Glimmer использовался для 73% геномов, для которых методы могли быть однозначно идентифицированы. ) Glimmer использовался DDBJ для повторного аннотирования всех бактериальных геномов в международных базах данных нуклеотидных последовательностей.[7] Эта группа также использует его для аннотирования вирусов.[8] Glimmer является частью конвейера бактериальных аннотаций в Национальном центре биотехнологической информации (NCBI),[9] который также поддерживает веб-сервер для Glimmer,[10] как и сайты в Германии,[11] Канада,.[12]
Согласно Google Scholar, по состоянию на начало 2011 года оригинальная статья Glimmer (Salzberg et al., 1998)[1] цитировался 581 раз, а статья Glimmer 2.0 (Delcher et al., 1999)[4] был процитирован 950 раз.
Рекомендации
- ^ а б c d е ж грамм час я Зальцберг, С. Л .; Delcher, A. L .; Kasif, S .; Уайт, О. (1998). «Идентификация микробных генов с использованием интерполированных марковских моделей». Исследования нуклеиновых кислот. 26 (2): 544–548. Дои:10.1093 / nar / 26.2.544. ЧВК 147303. PMID 9421513.
- ^ Зальцберг, С. Л .; Pertea, M .; Delcher, A. L .; Гарднер, М. Дж .; Теттелин, Х. (1999). «Интерполированные марковские модели для поиска эукариотических генов». Геномика. 59 (1): 24–31. CiteSeerX 10.1.1.126.431. Дои:10.1006 / geno.1999.5854. PMID 10395796.
- ^ «Центр вычислительной биологии». Университет Джона Хопкинса. Получено 23 марта 2013.
- ^ а б c d е Delcher, A .; Harmon, D .; Kasif, S .; Белый, O .; Зальцберг, С. (1999). «Улучшенная идентификация микробных генов с помощью GLIMMER». Исследования нуклеиновых кислот. 27 (23): 4636–4641. Дои:10.1093 / nar / 27.23.4636. ЧВК 148753. PMID 10556321.
- ^ а б c d е Delcher, A. L .; Братке, К. А .; Пауэрс, Э. С .; Зальцберг, С. Л. (2007). «Идентификация бактериальных генов и ДНК эндосимбионтов с помощью Glimmer». Биоинформатика. 23 (6): 673–679. Дои:10.1093 / биоинформатика / btm009. ЧВК 2387122. PMID 17237039.
- ^ Косуге, Т .; Abe, T .; Окидо, Т .; Tanaka, N .; Hirahata, M .; Maruyama, Y .; Mashima, J .; Томики, А .; Курокава, М .; Himeno, R .; Fukuchi, S .; Миядзаки, S .; Gojobori, T .; Tateno, Y .; Сугавара, Х. (2006). «Исследование и оценка возможных генов из 183 бактериальных штаммов с помощью общего протокола для идентификации новых генов: генный путь в прокариотном пространстве (GTPS)». ДНК исследования. 13 (6): 245–254. Дои:10.1093 / dnares / dsl014. PMID 17166861.
- ^ Sugawara, H .; Abe, T .; Gojobori, T .; Татено, Ю. (2007). «DDBJ работает над оценкой и классификацией бактериальных генов в INSDC». Исследования нуклеиновых кислот. 35 (Проблема с базой данных): D13 – D15. Дои:10.1093 / нар / gkl908. ЧВК 1669713. PMID 17108353.
- ^ Hirahata, M .; Abe, T .; Tanaka, N .; Kuwana, Y .; Shigemoto, Y .; Миядзаки, S .; Suzuki, Y .; Сугавара, Х. (2007). «Брокер информации о геноме для вирусов (GIB-V): база данных для сравнительного анализа геномов вирусов». Исследования нуклеиновых кислот. 35 (Проблема с базой данных): D339 – D342. Дои:10.1093 / нар / gkl1004. ЧВК 1781101. PMID 17158166.
- ^ "Система автоматической аннотации прокариотических геномов NCBI (PGAAP)". Центр биоинформатики и вычислительной биологии. Получено 23 марта 2012.
- ^ «Инструменты аннотации микробного генома». Центр биоинформатики и вычислительной биологии. Получено 23 марта 2012.
- ^ «ТиКо». Institut für Mikrobiologie und Genetik, Universität Göttingen. 2005-02-11. Получено 23 марта 2012.
- ^ «Система бактериальной аннотации BASys». Архивировано из оригинал 24 июля 2012 г.. Получено 23 марта 2012.
внешняя ссылка
- Домашняя страница Glimmer в CCB Университета Джона Хопкинса, с которого можно загрузить программное обеспечение.





![{ displaystyle operatorname {IMM_ {k} (S_ {x}) = Y_ {k} (S_ {x-1}) cdot P_ {k} (S_ {x}) + [1- {Y_ {k} (S_ {x-1})] cdot IMM_ {k-1} (S_ {x})}}}](https://wikimedia.org/api/rest_v1/media/math/render/svg/2d303028692d57e04cae6e304ca63cd7db9b1181)



![{ displaystyle operatorname {P_ {i} (S_ {x}) = P (s_ {x} | S_ {x, j}) = { frac {f (S_ {x, j})} { sum _ {b in {[acgt]}} operatorname {f (S_ {x, i}, b)}}}}}](https://wikimedia.org/api/rest_v1/media/math/render/svg/0ed6988a29b7c1f3de8c00efa5f2bd50e895dc06)












