Маркеры ДНК, связанные с сайтом рестрикции - Restriction site associated DNA markers
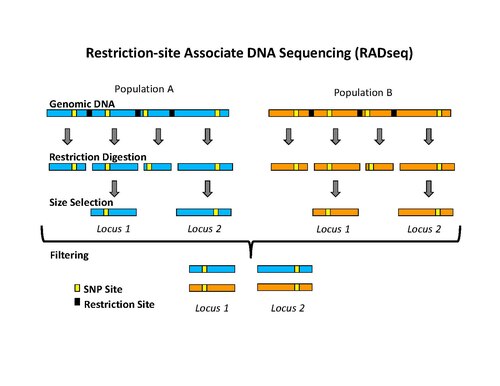
Маркеры ДНК, связанные с сайтом рестрикции (RAD) являются разновидностью генетический маркер которые полезны для сопоставления ассоциаций, QTL-отображение, популяционная генетика, экологическая генетика и эволюция. Использование маркеров RAD для генетического картирования часто называют картированием RAD. Важным аспектом маркеров и картирования RAD является процесс выделения тегов RAD, которые представляют собой последовательности ДНК, которые непосредственно фланкируют каждый экземпляр определенного сайта рестрикции рестрикционный фермент по всему геному.[1] После выделения меток RAD их можно использовать для идентификации и генотипирования полиморфизмов последовательностей ДНК в основном в виде однонуклеотидные полиморфизмы (SNP).[1] Полиморфизмы, которые идентифицируются и генотипируются путем выделения и анализа тегов RAD, называются маркерами RAD.
Изоляция тегов RAD
Использование фланкирующих последовательностей ДНК вокруг каждого сайта рестрикции является важным аспектом меток RAD.[1] Плотность меток RAD в геноме зависит от рестрикционного фермента, используемого в процессе выделения.[2] Существуют и другие методы маркеров сайтов рестрикции, например RFLP или же полиморфизм длины амплифицированного фрагмента (AFLP), которые используют полиморфизм длины фрагмента, вызванный различными сайтами рестрикции, для различения генетического полиморфизма. Использование фланкирующих последовательностей ДНК в методах RAD-меток называется методом сокращенного представления.[3]
Первоначальная процедура выделения меток RAD включала переваривание ДНК определенным рестрикционным ферментом, лигирование биотинилированный переходники на свесы, произвольно стрижка ДНК на фрагменты, намного меньшие, чем среднее расстояние между сайтами рестрикции, и выделение биотинилированных фрагментов с использованием стрептавидин бусы.[1] Эта процедура первоначально использовалась для изоляции тегов RAD для микрочип анализ.[1][4][5] Совсем недавно процедура изоляции тегов RAD была изменена для использования с высокопроизводительным секвенированием на Иллюмина платформа, преимущество которой заключается в значительном снижении количества необработанных ошибок и высокой пропускной способности.[2] Новая процедура включает в себя переваривание ДНК определенным рестрикционным ферментом (например: SbfI, NsiI,…), лигирование первого адаптера, называемого P1, к выступам, случайным образом разрезая ДНК на фрагменты, намного меньшие, чем среднее расстояние между сайтами рестрикции. подготовка обрезанных концов в тупые концы и лигирование второго адаптера (P2) и использование ПЦР для специфической амплификации фрагментов, содержащих оба адаптера. Важно отметить, что первый адаптер содержит штрих-код короткой последовательности ДНК, называемый MID (молекулярный идентификатор), который используется в качестве маркера для идентификации различных образцов ДНК, которые объединяются и секвенируются в одной реакции.[2][6] Использование высокопроизводительного секвенирования для анализа тегов RAD можно классифицировать как секвенирование с сокращенным представлением, которое включает, среди прочего, RADSeq (RAD-Sequencing).[3]
Обнаружение и генотипирование маркеров RAD
После выделения меток RAD их можно использовать для идентификации и генотипирования полиморфизмов последовательностей ДНК, таких как однонуклеотидные полиморфизмы (SNP).[1][2] Эти полиморфные сайты называют маркерами RAD. Самый эффективный способ найти теги RAD - это высокая пропускная способность. Секвенирование ДНК,[2][6] называется последовательностью тегов RAD, последовательностью RAD, RAD-Seq или RADSeq.
До разработки технологий высокопроизводительного секвенирования маркеры RAD были идентифицированы путем гибридизации тегов RAD с микрочипами.[1][4][5] Из-за низкой чувствительности микрочипов этот подход может обнаруживать либо полиморфизмы последовательностей ДНК, которые нарушают сайты рестрикции и приводят к отсутствию меток RAD, либо существенные полиморфизмы последовательностей ДНК, которые нарушают гибридизацию меток RAD. Следовательно, плотность генетических маркеров, которая может быть достигнута с помощью микрочипов, намного ниже, чем то, что возможно с помощью высокопроизводительного секвенирования ДНК.[7]
История
Маркеры RAD были сначала реализованы с использованием микрочипов, а затем адаптированы для NGS (Next-Generation-Sequencing).[7] Он был разработан совместно лабораториями Эрика Джонсона и Уильяма Креско в Орегонский университет примерно в 2006 г. Они подтвердили полезность маркеров RAD, определив контрольные точки рекомбинации в D. melanogaster и обнаружением QTL у трехиглой колюшки.[1]
В 2012 году ученые опубликовали модифицированный метод маркировки RAD под названием двойной дайджест RADseq.[8] Они добавили второй рестрикционный фермент и этап жесткого выбора размера ДНК для проведения недорогого генотипирования населения.
Источники
- ^ а б c d е ж грамм час Миллер MR; Dunham JP; Amores A; Креско WA; Джонсон EA (2007). «Быстрая и экономичная идентификация полиморфизма и генотипирование с использованием маркеров ДНК, ассоциированной с сайтами рестрикции (RAD)». Геномные исследования. 17 (2): 240–248. Дои:10.1101 / гр.5681207. ЧВК 1781356. PMID 17189378.
- ^ а б c d е Бэрд Н.А.; Etter PD; Этвуд Т.С.; Currey MC; Дрожь AL; Льюис З.А.; Selker EU; Креско WA; Джонсон EA (2008). «Быстрое открытие SNP и генетическое картирование с использованием секвенированных маркеров RAD». PLOS ONE. 3 (10): e3376. Дои:10.1371 / journal.pone.0003376.
- ^ а б Дэйви JW; Hohenlohe PA; Etter PD; Boone JQ; Catchen JM; Blaxter ML (2011). «Общегеномное открытие генетических маркеров и генотипирование с использованием секвенирования следующего поколения». Природа Обзоры Генетика. 12: 499–510. Дои:10.1038 / nrg3012. PMID 21681211.
- ^ а б Миллер MR; Этвуд Т.С.; Eames BF; Eberhart JK; Ян Ю.Л .; Postlethwait JH; Джонсон EA (2007). «Микромассивы маркеров RAD позволяют быстро картировать мутации рыбок данио». Геном Биол. 8 (6): R105. Дои:10.1186 / gb-2007-8-6-r105.
- ^ а б Льюис З.А.; Дрожь AL; Ребро жесткости N; Миллер MR; Джонсон EA; Селкер ЕС (2007). «Выявление высокой плотности маркеров ДНК, ассоциированных с сайтами рестрикции (RAD), для быстрого картирования мутированных локусов в Neurospora». Генетика. 177 (2): 1163–1171. Дои:10.1534 / генетика.107.078147. ЧВК 2034621. PMID 17660537.
- ^ а б Hohenlohe PA; Bassham S; Etter PD; Ребро жесткости N; Джонсон EA; Креско WA (2010). «Популяционная геномика параллельной адаптации трехиглой колюшки с использованием секвенированных RAD-тегов». PLoS Genetics. 6 (2): e1000862. Дои:10.1371 / journal.pgen.1000862. ЧВК 2829049. PMID 20195501.
- ^ а б Shendure J; Джи Х (2008). «Секвенирование ДНК нового поколения». Природа Биотехнологии. 26: 1135–1145. Дои:10.1038 / nbt1486. PMID 18846087.
- ^ Hohenlohe PA; Bassham S; Etter PD; Ребро жесткости N; Джонсон EA; Креско WA (2012). «Популяционная геномика параллельной адаптации трехслойной колюшки с использованием секвенированных меток RAD». PLOS ONE. 7: e37135. Дои:10.1371 / journal.pone.0037135. ЧВК 3365034. PMID 22675423.
